pecoでcdを快適にした|bashでもpeco
はじめに
pecoってなに?という方はこのへん↓をご参考に。
GitHub - peco/peco: Simplistic interactive filtering tool
ダウンロードは一番上の公式サイトから。
cdで遠くのディレクトリに移動するとき、いくらtabキーがあるとはいえ、長ったらしいパスを打ち込んで目的のディレクトリに一気に移動している人は少ないだろう。
かといって、一つ一つcd→ls→cdみたいに進んでいくのもめんどくさい。
という方にオススメのコマンドをpecoで作ってみた。
仕様
まずは↓を見ていただきたい。「sd」というのがそのコマンドだ。

基本動作は、
pecoで次々とディレクトリ選択して、目的のディレクトリまで一気に移動する
といった感じになる。
上のgifアニメでは、矢印キーでポチポチ選択しているが、もちろん検索して移動していくこともできる。
一番上の項目には常に今のフルパスを表示されるようになっており、それを選べばプロセスが完了する。
上から二番目の「../」を選択すると直上のディレクトリにも移動できる。
さらにオプションとして、①隠しディレクトリを表示する機能、②今いるディレクトリの中のファイルを表示する機能、③いつでもホームディレクトリに移動できる機能を追加した。詳しくは後述。
スクリプト
### .bashrcに記述 ###
function peco-cd {
local sw="1"
while [ "$sw" != "0" ]
do
if [ "$sw" = "1" ];then
local list=$(echo -e "---$PWD\n../\n$( ls -F | grep / )\n---Show hidden directory\n---Show files, $(echo $(ls -F | grep -v / ))\n---HOME DIRECTORY")
elif [ "$sw" = "2" ];then
local list=$(echo -e "---$PWD\n$( ls -a -F | grep / | sed 1d )\n---Hide hidden directory\n---Show files, $(echo $(ls -F | grep -v / ))\n---HOME DIRECTORY")
else
local list=$(echo -e "---BACK\n$( ls -F | grep -v / )")
fi
local slct=$(echo -e "$list" | peco )
if [ "$slct" = "---$PWD" ];then
local sw="0"
elif [ "$slct" = "---Hide hidden directory" ];then
local sw="1"
elif [ "$slct" = "---Show hidden directory" ];then
local sw="2"
elif [ "$slct" = "---Show files, $(echo $(ls -F | grep -v / ))" ];then
local sw=$(($sw+2))
elif [ "$slct" = "---HOME DIRECTORY" ];then
cd "$HOME"
elif [[ "$slct" =~ / ]];then
cd "$slct"
elif [ "$slct" = "" ];then
:
else
local sw=$(($sw-2))
fi
done
}
alias sd='peco-cd'
以上を.bashrcとかに書き込んで、
source .bashrc
で読み込めば使用できるようになり、
sd
で実行される。Select Directoryの略のつもり。
ショートカットが気に食わない方は適宜改変してください。
オプション
①隠しディレクトリを表示する機能
下から3番目の「---Show hidden directory」を選択することで、隠しディレクトリを表示するモードに移行する。
デフォルトでは隠しディレクトリは表示されないようになっているので、隠しディレクトリに移動したいときはこの項目を選択する必要がある。
隠しディレクトリが邪魔になったら「---Hide hidden directory」を選択すれば、再び隠してくれる仕様です。
②今いるディレクトリの中のファイルを表示する機能
下から2番目の項目に、今いるディレクトリのファイルを一行で表示するようにした。
さらに、この項目を選択すると、一覧にしてpecoで表示してくれるようになる。
ちなみにファイル一覧を表示しているときは何を選択しても、前の画面に戻るだけである。
③いつでもホームディレクトリに移動できる機能
一番下には「---HOME DIRECTORY」という項目を設けて、どこにいても一気にホームディレクトリに移動できるようにした。
ホームディレクトリ移動後もプロセスは終了しないので、ホーム直下のディレクトリなんかは一回ホームディレクトリを経由すると楽に移動できる。
おわりに
オプションコマンドをpecoの選択肢の中に組み込んだため、検索に引っかかってしまうという欠点がある。
私は基本的に矢印で移動しているので、あまり問題にならないが、ガンガン検索して移動していく人には邪魔になるかもしれない。
あと場合によっては、ディレクトリ移動後、一覧が表示されるまで少しラグが生じることがある。
もっと改善できそうなら、ご報告ください。
ポスターデザインの配色を考える|基本編
はじめに
学会発表用のポスターを作るとき、色使いに気を使っている人はどれくらいいるだろうか。
正直、適当に配色してもまあまあ伝わるポスターが作れる。
それよりも文字や図の配置、フォントなどに気を配った方が伝わるポスターになる。
人に伝えるという目的のために配色が担う役割は、あまり多くない。
ただ私の経験上、世の中には色使いで損しているポスターが結構あるように思う。
配色の役割を正確に理解することで、そのような失敗をしなくなるだけでなく、
伝えるポスターを作る上で配色を武器にすることが出来る。
私が以前、ポスターの配色についてネットで調べてみたとき、
わかったのは「赤色は情熱的」とか「紫色は高貴」とか、そういうことばっかりだった。
そんな本当かどうかもわからないようなことは学会ポスターを作る上ではわりとどうでもよくて、
私が知りたかったのは、どのような配色だと主張を効率よく伝えられるか、ということである。
その後、いろいろと自分で試行錯誤した結果、いろんなことに気がついたので、このページにまとめてみた。
ちなみに、全体的なポスターデザインについては、
学会発表用ポスターを美しくするためのコツ - マクロ生物学徒の備忘録
こちらを参考にしてもらえれば。
基本的なコンセプトはこちらと共通している。
配色の役割とは
まず、テンプレートとしてこんなポスターを用意した。


※内容は適当です。
いずれも無彩色で作ってみた。
配置などはきれいに揃えたつもりなのでそこそこ「伝わる」とは思う。
一方で、これだけ配置やフォントにこだわっても、わかりにくい点や問題点が少なからずある。
そこを指摘していくことで、「ポスターデザインにおける配色の役割」を考えていく。
問題点①「どこが重要なのかがわからない」
みなさんは、学会でポスターをどのように見ていくだろうか。
私の場合は、
・タイトルを見て、テーマを把握する。
・テーマに興味がなくとも、10秒ほど掛けておおまかな主張を読み取ろうとする。
→10秒で理解できないときは…
・テーマに興味がないなら諦めて次に行く。
・テーマに興味があるならあと1分ほどかけて読みこむ。
・主張が面白ければ、覚えておいて発表者に話を聞く。
という感じだが、どうだろう。
別の見方をする人も多いのかもしれないが、
いずれにせよ、短時間で大まかな主張が伝わるかはとても重要だと思う。
先程のポスターを見てみよう。
短時間で内容を把握したい場合、読む場所を取捨選択する必要があるが、
どこを読めばいいかわかるだろうか?
そう、太字のところをみればいい。私もそのように作ったつもりだ。
しかし「太字のところを見ればいい」と理解するのに何秒かかるだろうか?
察しのいい人で3秒、普通は5秒程度かかるんじゃないだろうか。
この「読むべき場所はどこか」を把握するために掛かる時間は、見る人にとっては無駄な時間である。
あまりにも時間がかかりそうな場合、忙しい人は読む気をなくしてしまうかもしれない(私もそうだ)。
よって、発表者はこの時間を極限にまで減らす努力をすべきである。
そこで、次のように改善してみた。


このポスターなら、誰がどう見ても「赤字を読めばいい」と理解できるだろう。
しかも、一瞬で。
つまり、
読んでほしい場所を強調する、
というのが配色の一番大きな役割と言えるだろう。
問題点②「見出しがわかりにくい」
このポスターでは各項目の左上に見出しがついている。
それはいい。
一方で、左下の結果の項目に注目してほしい。
小見出しがついているが、この小見出しと本文の区別が少しつきにくい。
小見出しがわかりにくいと、「結果が2つある」ということを把握するのに少し時間が掛かるということに繋がる。
次のように改善してみた。


大見出しを濃紺に、小見出しを紺にしてみた。
こうすると、見出しがわかりやすくなり、内容も把握しやすくなるだろう。
見出しと本文の区別をつける、
というのも、配色の役割の1つだろう。
問題点③「全体的に地味」
ポスターを見るとき、まずポスター会場を適当にふらふらして、なんとなく気になるポスターだけ読んでいくという人もいるだろう。
そういう意味では、目立つポスターというのは有利だといえる。
また、目立つ配色は印象に残りやすい。
もちろん内容が伴っていないとだめだが、ポスター賞を狙う上でも、目立つというのは戦略の1つだろう。
そこで、こんな感じにしてみた。

テーマ色として水色を採用した。
印象として、随分明るくなったと思う。
周りのポスターにもよるが、そこそこ目立つんじゃないかと思う。
ちなみに、見出しの色もテーマ色に合わせて紺色から青色にしてみた。
見出しは、本文と区別がつけば十分なので、テーマ色と同じでもよい。
むしろ、同じ方が統一感があって良いかもしれない。
そのあたりは後述の「配色のコツ」にて考える。
テーマ色を何色にするかは、センスが問われるところだ。
この辺は応用編(近日公開予定)も参考にしてもらえれば。
配色のコツ
ここまで、ポスターデザインにおける配色には
① 読んでほしい場所を強調する
② 見出しと本文の区別をつける
③ ポスター全体を目立たせる
というような役割があることを指摘してきた。
こうした配色の役割を把握した上で、配色のコツを考えていく。
とにかく強調を殺さない
上述の①〜③の役割には、明確な優先順位が存在する。
内容を効率よく伝えるという目的のためには、なんといっても、①読んでほしい場所を強調するというのが一番大切である。
たとえば、以下の例を見てみよう。


テーマ色、強調色は先程と変わらず、見出しの色をオレンジ色にしてみた。
②見出しと本文の区別をつける、という項目はこれでも達成されている。
しかし、①はどうだろう。
見出しの色と強調色がともに赤系統で似ているため、若干、強調が弱くなっている。
そのため「赤字を読めばいい」というのが把握しにくくなってしまった。
失敗である。
以下の例も見てみよう。


これも、強調がすこしだけわかりにくくなっている。
また、見出しが変に目立ちすぎている気もする。
いや、強調わかるじゃん!って思うかもしれないが、




こうして並べてみると、上よりも、下の方が強調部分がはっきり浮き出ており、
読むべきところが一瞬で把握できるのがわかるだろう。
どうしても、テーマ色に赤色を使いたいなら、


こういう感じはどうだろう。
青色や緑色はあまり強調に向かない色ではあるので、ちょっとわかりにくいかもしれない。
そのへんを改善したければ、以下のようにする。


赤色は強い色なので、少し鈍くして印象を弱くし、
逆に強調色の青色をビビットにして印象を強くした。
色数は少ない方がいい
これも強調を殺さないというのとほとんど同じ話だが、色数は少ない方がいい。
色数が多いと煩雑になって、強調がわからなくなりやすい。
以下の2つを見比べてもらえればわかると思う。
色数多い


色数少ない


色数が少ないの方が圧倒的に強調がわかりやすい。
まとめられる色はできるだけまとめて、彩度や明度で区別をつけると良い。
…しかし、色数が多くても伝わりやすいポスターを作ることも不可能ではない。
冒頭で、色数が多くなることで強調がわからなくなるのが問題だと述べた。
逆に言えば、強調色を邪魔しない限り、色数を多くしても問題はない。
この辺は応用編(近日公開予定)で述べる。
まとめ
配色には以下の役割がある。
① 読んでほしい場所を強調する
② 見出しと本文の区別をつける
③ ポスター全体を目立たせる
このうち、特に①に関してはとくに重要で、
・強調色は強調したくないところには使わないようにする
・強調色と同系統の色もできるかぎり避ける
・色数は少なめにする
ことを心がけると強調がより明確になって、自分の主張をもっと効率良く伝えることができる。
Wineを使ってArlequinを起動する|OS X ElCapitan (10.11.6)
はじめに
集団遺伝学ツールとして有名なArlequin(アルルカン)。
HW平衡からの逸脱を調べたり、遺伝構造の階層を見たりと、何かと重宝する。
しかし、Windows専用というのが、なかなかめんどくさい。
そこで、WineをインストールしてMac上でWindowsソフトを実行できるようにして、Arlequinを起動させてみた。
この項では、
Wineのインストールと設定
Arlequinのダウンロード
Automatorを使ったアプリ化(おまけ)
について解説する。
Wineのインストールと設定
今まで、MacでWin用ソフトを立ち上げるWineというアプリがあるのは知っていたが、Wineとググると色んなものが出てきてどれが何なのかイマイチよくわからなかった。
そもそもWineとは、Windowsソフトを別のOSで起動させるためのツールの総称(?)のようなものなのかも。
今回は、このサイトのWine.appを使う。
WineBottler | Run Windows-based Programs on a Mac
このサイトでは、「WineBottler」と「Wine.app」の2つのソフトを提供している。
Wine.appは、Windowsソフトを立ち上げるためのアプリであり、
WineBottlerは、アプリケーションをインストールしたりできる。
Arlequinに限っていえば、WineBottlerは全く必要ないが、お好みで。
ちなみにWineBottlerをダウンロードすればWine.appも同梱されているので、わざわざ2つをダウンロードする必要はない。
ここでは、Wine.app をダウンロードする。
WineBottler | Downloads of WineBottler and Wine.app
上記サイトを開き、下にスクロール。
 この赤枠部分をクリック(ElCapitanの場合)。
この赤枠部分をクリック(ElCapitanの場合)。
こんなダイアログが出てくるのでDownloadをクリック。
広告サイトに飛ぶので、スルーして5秒ほど待つ。

すると、右上が「SKIP AD」となるので、クリック。
しばらく待つとWine_1.8-rc4.dmgのダウンロードが始まるはず。
ダウンロードが終わったら、Wine_1.8-rc4.dmgを起動する。

こんなのが出てくるのでWineのアイコンをドラッグしてApplicationsにコピーする。
これでインストール完了。
次に設定をおこなう。
Launchpadなどから今インストールしたWineを起動する。

画面右上にWineのアイコンが表示されるはず(画像赤枠)。
このアイコンをクリックし、「Change Prefix...」を選択する。
すると、どこに仮想的なWindows環境を作るかを設定できる。
好きな場所を設定しよう。
複数用意してソフトごとに使い分けることもできるが、あまり意味はない。
使わないやつは消してよい。
作ったフォルダをFinderで開いてみると、drive_cというフォルダが確認できる。
仮想的にCドライブを作って、Windowsのような環境を作っているようだ。
Arlequinのダウンロード
ここから、「winarl35.zip」をダウンロードする。
ダウンロードしたファイルを好きな場所に展開する。
できたWinArl35フォルダを開くと、「WinArl35.exe」というファイルがあるはず。
Wineが起動している状態(=右上にWineアイコンがある状態)で「WinArl35.exe」をダブルクリックすると、Arlequinが起動するはず。
起動までに時間がかかるのはご愛嬌ということで。
(おまけ)Automatorを使ったアプリ化
これでMacでもArlequinを使えるようになったわけだが、
Wineを立ち上げて、WinArl35フォルダを開いて、「WinArl35.exe」をダブルクリックする、というのを毎回やるのはちょっとめんどくさい。
普通のMacアプリのようにLaunchpadからワンクリックで起動できないものか…。
ということで、Mac標準アプリのAutomatorを使って、ArlequinをLaunchpadからワンクリックで開けるようにする。
まず、Automatorを起動する。
意外と知られていないがMacに標準装備されているアプリなので、必ずあるはず。

起動して、新規書類を選択するとこういう画面になるはず。
「アプリケーション」をクリックして「選択」。

「ユーティリティ」→「アプリケーションを起動」を右のスペースにドラッグ。
さらに「シェルスクリプトを実行」もドラッグ。

こんなかんじ。
「アプリケーションを起動」の部分のプルダウンから「Wine」を選択する。
さらに、シェルスクリプトを実行の枠内のcatを消して、以下の文を追加する。
sleep 1
open ~/○○/△△/WinArl35/WinArl35.exe※環境によっては一行目はなくてもいけるかも。
※openの後ろはWinArl35を解凍した場所を指定してください。

完成形はこんな感じ。
できたら、左上の「ファイル」から「保存」。
「Arlequin」でも「WinArl35」でも好きな名前にしてApplicationsに保存する。
すると、Launchpadに表示されるようになるはず。
これで、LaunchpadからワンクリックでArlequinを起動できるようになった。
研究者のためのAlfredの設定
はじめに
Alfredとは、言わずと知れたランチャーアプリ。
アプリの起動はもちろん、ファイルやメールなどの検索もこなしてしまう優れものだ。
Alfredを入れていないMacはMacではないと言い切る人もいるほど、定番アプリとして名高い。
ダウンロードは↓からできる。
Alfred - Productivity App for Mac OS X
基本的な使い方は、
Mac - ランチャーアプリ「Alfred」の基本的な使い方 - PC設定のカルマ
Alfredの基本的な使い方を一通り網羅して覚える(前編) | ごりゅご.com
このへんを見ればだいたいわかるはず。
この項では、研究者ならみんな使いそうな設定と、バイオ系の研究者にとって便利な設定を紹介する。
Google Scholarで論文検索
Alfredを起動し、preferenceを開く。
FeaturesのWeb searchの項目を開く。
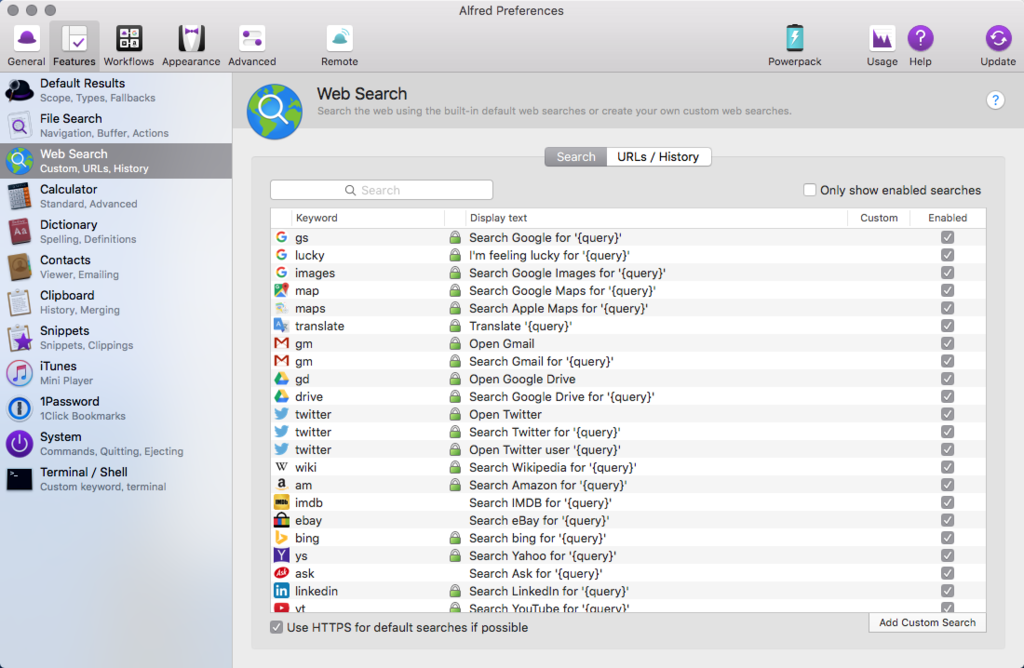
こんな画面になるはず。
右下の「Add Custom Seach」をクリック。
Search URLの項目に
http://scholar.google.com/scholar?q={query}
と入力する。
タイトルやキーワードは適当にきめればよい。
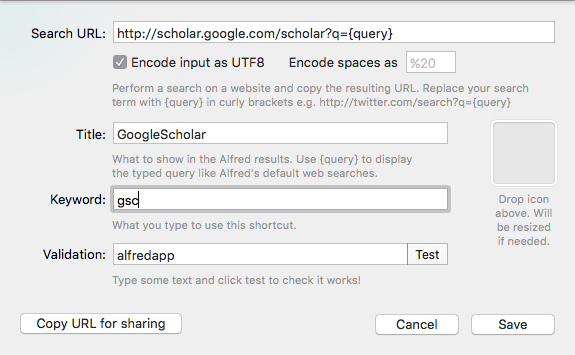
例えばこんな感じ。
これで、Alfredを起動した後、
gsc 検索ワード
※間は半角スペース
と打ち込めばGoogle Scholarで検索してくれる。便利だ。
PubMedで論文検索
上記のGoogleScholarとほぼ同じ手順で設定する。
Search URLの項目には、
http://www.ncbi.nlm.nih.gov/pubmed/?term={query}
と入力する。
キーワードは適当に。「pm」とか。
ALCで英語翻訳
これがとても便利。
英語の論文を読んでいるときに頻繁に使用する。
やはり上記のGoogleScholarとほぼ同じ手順で設定する。
Search URLの項目には、
http://eow.alc.co.jp/{query}/
と入力する。
キーワードはなんでもいいが、頻繁に利用するので短めがオススメ。
自分は「t」の一文字に設定している。
NCBIでキーワード検索する
バイオ系研究者なら頻繁に使うであろうデータベースのNCBI。
これも設定してしまおう。
AllDataBaseで検索するには、
http://www.ncbi.nlm.nih.gov/gquery/?term={query}
をSearch URLの項目に入力すればいい。
Geneで検索するには、
http://www.ncbi.nlm.nih.gov/gene/?term={query}
Proteinで検索するには、
http://www.ncbi.nlm.nih.gov/protein/?term={query}
をそれぞれ入力する。
自分は、キーワードをそれぞれ「ncbi」「ncbig」「ncbip」に設定している。
BLASTで検索…は無理っぽい?
こんな感じでBlast検索もできるんじゃないかと思ったが上手くいかなかった。
トップページにすぐアクセスできるようにするくらいか。
上手くいった人は教えてください。
学会発表用ポスターを美しくするためのコツ
はじめに
まず、こちらのサイトを熟読すべし。
「伝える」という目的のためには、このサイトに書いてあることがすべてだと思う。
まだ見たことがない人は今すぐ読むべし。本当に。まじで。
よくまとまっており、ひととおり目を通すのに1時間もかからないので、まずそちらから。
「伝わるデザイン」では、センスのない人でも人に伝えるためのルールが載っている。
この項では、伝えるという目的を前提にさらに美しく見せるためにはどうすればいいかを考える。
「人に伝える」という目的に比べ、「美しく見せる」という目的は非常に曖昧なものなので、私の主観を多分に含むことになる。ご容赦を。
ここでは、以下のコンセプトに則ったデザインを「美しいデザイン」と定義する。
・伝わるデザインであること
…伝わるデザインのコンセプトと矛盾しないデザインを目指す。
・強調は必要最小限かつ効果的に
…重要な情報以外を控えめにし、強調したい部分を効果的に見せるデザインを目指す。
・フラットデザインを重視する
…不必要な装飾を排し、シンプルで上品なデザインを目指す。
これらのコンセプトのもと、美しいポスター作りのコツをまとめてみた。
「伝わるデザイン」ともやや重複するところもあるし、かなり個人的な意見が入っているところもある。
適当に取捨選択してもらえれば。
こんなポスターはダサい
次のポスターをみてみよう。

※内容は適当です
こんな感じのポスターを学会でみかけたことのある人も多いだろう。
人によるとは思うが、お世辞にもかっこいいとは言えない。というかダサい。
これを元に、悪いところを指摘し、美しいデザインに修正していく。
1. フラットデザインを導入しよう
フラットデザインとは、近年、webやアプリのデザインで主流のコンセプトである。
詳しくはググってほしいが、かつて主流だったデザインは、現実の電子機器のメタリックな部分を模倣して、グラデーションがかかっていたり、影がついていたりした。
昔はこういうデザインが目を引き、未来的な印象を与えていたが、今では逆に古臭くなりつつある。
そんななか登場したフラットデザインは、文字通り平面的なデザインであり、派手さはないものの、ムダな装飾がなくシンプルで洗練されたデザインであるといえる。
たとえば、かつてのGoogleのロゴは

こんな感じだったが、今は、

こんな感じである。
Windowsのロゴも昔は

こうだったが、今は、

こんな感じ。これがフラットデザインである。
どうだろうか。
昔の方が好きだった、という人もいるだろうが、これが時代の流れというもの。
つまり、今の価値観から言えば、影やグラデーションはダサい、ということだ。
そもそも、影もグラデーションも、何の情報ももってないのに、わざわざ使用する必要がない。
過度な装飾は、ノイズにしかならず、重要な情報を見づらくしてしまいかねない。
では、先ほどのポスターから影とグラデーションをなくしてみよう。

こんな感じ、うーん、まだダサい。
2. 枠で囲わないようにしよう
このポスターは枠の印象が強すぎる。
一番外の大枠、各項目の中枠、さらに強調や図の小枠と、最大で3段階になっており、しかもすべて線も太くて色も濃い。
当たり前だが、枠はなんの情報ももっていない。必要最小限にすべき。
世の中にあふれる学会ポスターはこの点を踏まえるだけでずっと見やすくなるはず。
では、枠をなくしてみる。

枠をなくすと項目ごとのまとまりが見づらくなるため背景の色を少し濃くした。
どうだろう、かなりスマートになったような気が。
3. 角丸四角はダサい
これは正直、好みのわかれるところかもしれないが、角丸はダサい。
百歩譲って角丸を使うにしても、Rを小さめにして、下のように文字の端とそろえるべきだ。


これをふまえて、ポスターを修正してみる。
最初はRを小さくした例から。

まあ、これでも悪くはない。
次に、丸角をやめてただの四角にした例。

個人的にはこっちの方が好みだ。
4. 見出しを目立たせすぎない
ここでいう見出しとは、「Introduction」とか「Result」とかのこと。
ほとんどのポスターは「背景」「目的」「手法」「結果」「考察」で構成されている。
その順番に則っていて、かつ、逆N字型かZ型など一般的な構成である場合、見出しがなくてもそんなに困らないはず。
見出しの役割は、その項目に何が書いてあるかを示すものであり、内容とは関係ない。
見出し自体には情報がないのだから目立たせなくていいというわけ。
今回の例では、一番重要な部分と見出しがほぼ同じレベルで強調されている。
色付き背景というのもダサい。ここを修正する。

テーマカラーのピンクにしてみた。
このくらいでいいだろう。
ところで、見出しの隣に大きめなデッドスペースがある。
デザイン的には、このスペースは空白のままでもスッキリしてて良いかもしれない。
しかし、ここはわりと視線が行きやすいところなので、もったいない気もする。
とくに、左上のイントロの横はめちゃくちゃ視線が集まるところなので、キーワードやパワーワードを盛り込んでキャッチーな見出しにすると効果的だろう。
その他の見出しの横にも「一言まとめ」のようなものがあると親切だ。

次からは色についてみていく。
5. テーマカラーと強調色を使い分ける
テーマカラーとは、そのポスターの全体の印象を決める色である。
強調色とは、自分の主張したいところ、とりわけ重要なところに使用する色のことだ。
重要なのは、次の2点。
・テーマカラーと強調色を同系統の色にしないこと
・強調色はできる限り最小限にすること
とくに、後者はあまり意識されないことが多いので注意。

強調色はそのままで、テーマカラーを緑にした。
また、強調したいところ以外はテーマカラーと同系統の色にした。
ここまでくると、逆に強調がウザくなってくる。

この程度でも強調の役割は十分に果たしている。
こっちの方が上品に見える。
配色は自由度が高い分、悩むことも多いかもしれない。
配色について、もっと知りたい方は以下のページも参考にしてもらえると。
ポスターデザインの配色を考える|基本編 - マクロ生物学徒の備忘録
おわりに
いろいろといじったら、細かいところがずれてきたので最後に修正する。
左端揃えを徹底し、区切れの線の位置を微調整した。

すっきりと上品なポスターに仕上がった。
ちなみに、こういう感じのデザインで実際に学会に出たことがある。
まわりのポスターがごちゃっとしたものが多かったのもあって、こういうスッキリしたデザインは逆に目立っていた。
これくらいのデザインがごろごろいるような学会(進化学会とか)では、もう一工夫あった方が良いかもしれない。そのあたりはまた今度まとめるかも。
もし、インパクトを付けたければ色を派手にしたり、アイキャッチを大きくしたりするといい。

こんな感じとかどうだろう。ちょっと色が下品かもしれないが。
アイキャッチの色を変えてみるのもあり。
以上、いろいろ書いたが、結局は好みによるところが大きい。
参考にしつつ、自分好みのデザインを見つけてもらえれば。
pyRADの使い方
はじめに
pyRADって何?どうやってインストールするの?って方はこちら↓
pyRADのインストール|OS X ElCapitan (10.11.6) - マクロ生物学徒の備忘録
ここではインストールが済んでいる前提で書きます。
pyRADの使い方は公式サイトのチュートリアルにわりとわかりやすく書いてある。
読めばだいたいわかるはず。
解釈に自信がないところもあるので、間違ってたらご指摘ください。
(間違っていても責任はとれませんのであしからず。)
大まかな流れは、インプットファイルを用意して、pyRADでパラメータファイルを作成し、パラメータファイルをテキストエディタで改変し、pyRADを実行する、という感じ。
この項では、
・インプットファイルについて
・パラメータファイルの作り方
・パラメータの各項目について
・pyRADの実行
・パラメータをいじってみた印象
について触れていく。
インプットファイルについて
fastq形式で、
・fastqファイルが個体ごとにわかれている状態
・fastqファイルが1つにまとまっており、個体識別用のバーコードが付いている状態
・圧縮されて〇〇.fastq.gzとなっている状態
のどれでもできる。
ちなみに、次世代シーケンサから返ってきたファイルをそのまま使ってもいいが、FASTX-toolkitなどを使って、処理しておいた方がSNPが多くとれてくる。そんな気がする。
FASTX-toolkitのインストール|OS X ElCapitan (10.11.6) - マクロ生物学徒の備忘録
一般に、次世代シーケンサが出力するデータは、リードの最後の方のクオリティが低くなる傾向がある。
FASTX-toolkitを使うと、後ろ側からクオリティが低い部分を削る処理が可能なので、これだけでもやっておくのがオススメ。
パラメータファイルの作り方
pythonが立ち上がっているとして、
cd ~/Desktop/MIG/pyrad
python ~/Analysis/pyrad/pyrad/pyRAD.py -n ##pyRADをインストールしたディレクトリを指定する→params.txtが作られるはず。
このparams.txtを好きなテキストエディタで開くと、
==** parameter inputs for pyRAD version 3.0.66 **======================== affected step ==
./ ## 1. Working directory (all)
./*.fastq.gz ## 2. Loc. of non-demultiplexed files (if not line 18) (s1)
./*.barcodes ## 3. Loc. of barcode file (if not line 18) (s1)
vsearch ## 4. command (or path) to call vsearch (or usearch) (s3,s6)
muscle ## 5. command (or path) to call muscle (s3,s7)
TGCAG ## 6. Restriction overhang (e.g., C|TGCAG -> TGCAG) (s1,s2)
2 ## 7. N processors (parallel) (all)
6 ## 8. Mindepth: min coverage for a cluster (s4,s5)
4 ## 9. NQual: max # sites with qual < 20 (or see line 20)(s2)
.88 ## 10. Wclust: clustering threshold as a decimal (s3,s6)
rad ## 11. Datatype: rad,gbs,pairgbs,pairddrad,(others:see docs)(all)
4 ## 12. MinCov: min samples in a final locus (s7)
3 ## 13. MaxSH: max inds with shared hetero site (s7)
c88d6m4p3 ## 14. Prefix name for final output (no spaces) (s7)
==== optional params below this line =================================== affected step ==
## 15.opt.: select subset (prefix* only selector) (s2-s7)
## 16.opt.: add-on (outgroup) taxa (list or prefix*) (s6,s7)
## 17.opt.: exclude taxa (list or prefix*) (s7)
## 18.opt.: loc. of de-multiplexed data (s2)
## 19.opt.: maxM: N mismatches in barcodes (def= 1) (s1)
## 20.opt.: phred Qscore offset (def= 33) (s2)
## 21.opt.: filter: def=0=NQual 1=NQual+adapters. 2=strict (s2)
## 22.opt.: a priori E,H (def= 0.001,0.01, if not estimated) (s5)
## 23.opt.: maxN: max Ns in a cons seq (def=5) (s5)
## 24.opt.: maxH: max heterozyg. sites in cons seq (def=5) (s5)
## 25.opt.: ploidy: max alleles in cons seq (def=2;see docs) (s4,s5)
## 26.opt.: maxSNPs: (def=100). Paired (def=100,100) (s7)
## 27.opt.: maxIndels: within-clust,across-clust (def. 3,99) (s3,s7)
## 28.opt.: random number seed (def. 112233) (s3,s6,s7)
## 29.opt.: trim overhang left,right on final loci, def(0,0) (s7)
## 30.opt.: output formats: p,n,a,s,v,u,t,m,k,g,* (see docs) (s7)
## 31.opt.: maj. base call at depth>x<mindepth (def.x=mindepth) (s5)
## 32.opt.: keep trimmed reads (def=0). Enter min length. (s2)
## 33.opt.: max stack size (int), def= max(500,mean+2*SD) (s3)
## 34.opt.: minDerep: exclude dereps with <= N copies, def=1 (s3)
## 35.opt.: use hierarchical clustering (def.=0, 1=yes) (s6)
## 36.opt.: repeat masking (def.=1='dust' method, 0=no) (s3,s6)
## 37.opt.: vsearch max threads per job (def.=6; see docs) (s3,s6)
==== optional: list group/clade assignments below this line (see docs) ==================こうなっているはず。
##14までは必須で、それ以降は任意。
集団の情報を入力するには「optional: list group/clade assignments below this line」以下に記述する。
パラメータの各項目について
まだよく理解しきれていないところもある。
そのへんはご勘弁を。
いろいろ自分でいじってみてどうなったかは、あとの章で述べる。
## 1. Working directory
作業ディレクトリを指定する。
ここにアウトプットファイルが出力される。
相対パス、絶対パスのどちらでもいい。
## 2. Loc. of non-demultiplexed files (if not line 18)
インプットファイルのパスを指定。
複数のファイルを使用したいときは、*(ワイルドカード)も使える。
ただし、バーコードなどがついたままのrawデータ(.fastq / .fastq.gz)に限る。
バーコードが取れたあとのファイルを使用したい場合は、ここは空欄にする。
## 3. Loc. of barcode file (if not line 18)
バーコードファイルのパスを指定。
バーコードが取れたあとのファイルを使用したい場合は、ここも空欄にする。
## 4. command (or path) to call vsearch (or usearch)
vsearchのコマンドを指定する。
パスが通っている場合は
vsearch
と書くだけ。
相対パスか絶対パスを記述することもできる。
## 5. command (or path) to call muscle
muscleのコマンドを指定する。
muscleはダウンロードしたままの状態だと、
muscle3.8.31_i86darwin64
みたいな感じになっている。
muscle3.8.31_i86darwin64 → muscle にリネームしていれば、
muscle
だけでよい。
相対パスか絶対パスを記述することもできる。
## 6. Restriction overhang (e.g., C|TGCAG -> TGCAG)
RAD-seqの場合、使用した制限酵素サイトを指定する。
すでに制限酵素サイトがfastqファイルから除かれいるなら空欄でいいはず。
MIG-seqなど、制限酵素を使用していない場合も空欄でいい。
## 7. N processors (parallel)
どれだけパラレルに作業をするか。
2にすると2つ同時進行、3にすると3つ同時進行で作業をする。
多けりゃいいってもんではない。
使用するコンピューターのコア数と同じにするといいとか。
## 8. Mindepth: min coverage for a cluster
リードを採用する最低カバレッジ数。
数字を大きくすれば、質の高くてSNP数の少ないデータに、
小さくすれば、質が低くてSNP数の多いデータになる。
個人的には最低10くらいはほしい気がする。
## 9. NQual: max # sites with qual < 20 (or see line 20)
1つのリードにつき、シーケンスできていないサイト(N)をいくつまで許すか。
short readの場合と、long readの場合で、適正値が違う。
FASTX-toolkitで後ろから削る処理をしていない場合、この処理で大幅に使えるリードが減ってしまう。たぶん。
## 10. Wclust: clustering threshold as a decimal
何%一致した配列をクラスタリングするか。
例えば、90%にしたい場合
.90
という感じに記述する。
## 11. Datatype: rad,gbs,pairgbs,pairddrad,(others:see docs)
rad, ddrad, gbs, pairddrad, pairgbs, mergeが使用できるらしい。
どう違うかはよく知らない。
## 12. MinCov: min samples in a final locus
何個体以上で共通して読めたリードを採用するか。
数字を大きくすれば、質の高くてSNP数の少ないデータに、
小さくすれば、質が低くてSNP数の多いデータになる。
## 13. MaxSH: max inds with shared hetero site
これが未だによくわからない。
ヘテロの個体があまりにも多い場合、その座位はSNPじゃなくてパラログが同じクラスターにまとめられているだけという可能性が高い。
何個体以上でパラログに認定するか、という閾値をここで設定する、かな?たぶん。
## 14. Prefix name for final output (no spaces)
アウトプットファイルの名前。
なんでもよい。
以下はオプションの設定。
だいたい空欄でいいはず。
めぼしい項目だけ解説する。
## 18.opt.: loc. of de-multiplexed data
バーコードが取れたあとの個体ごとに分かれたfastqファイルを使用する場合、ここで指定する。
## 2と同様、ワイルドカードが使える。
## 20.opt.: phred Qscore offset (def= 33)
クオリティフィルターの閾値。
## 30.opt.: output formats: p,n,a,s,v,u,t,m,k,g,* (see docs)
どのアウトプットファイルを出力するか。
とりあえず「*」としておけば全部出力してくれるのでオススメ。
最後に集団の情報を記述する。
例えば、インプットファイルが
aaa001.fastq aaa002.fastq …
bbb001.fastq bbb002.fastq …
ccc001.fastq ccc002.fastq …
※aaa、bbb、cccは集団の名前
みたいなっている場合、次のようにする。
==== optional: list group/clade assignments below this line (see docs) ==================
1 4 aaa*
2 4 bbb*
3 4 ccc*一列目の数字は集団の番号。
集団の名前は入れられず、123とかの数字か、ABCとかのアルファベットのみ使用できるらしい。
二列目の数字はよくわからない。
これ以下の個体数しか得られなかった集団は放棄するってことかな?
三列目にファイル名を記入する。
例のようにワイルドカードを使ってもいいし、カンマ(,)区切りで並べていっても良い。
また、各列はタブで区切る。
pyRADの実行
pythonが立ち上がっている前提。
cd ~/Desktop/MIG/pyrad
python ~/Analysis/pyrad/pyrad/pyRAD.py -p params.txt ##pyRADをインストールしたディレクトリを指定するこれで動くはず。
コンピューターの性能やデータサイズにもよるが、普通のパソコンでやる場合、1〜3日くらいかかる。
ちなみに、pyRADは7つのステップに分かれている。
params.txtの(s1)、(s3,s6)、(all)といった表記はそのパラメータがどのステップに関わっているかを示したものである。
ステップ7に関わるパラメータだけをいじってやり直したいときは
-s 7
というオプションを追加するとステップ7だけやり直すことができる。便利。
パラメータをいじってみた印象
SNP数を増やしたかったり、質を上げたかったりする場合、いじれる項目は、
リードを採用する最低カバレッジ数(## 8)
何個体以上で共通して読めたリードを採用するか(##12)
がメインといったところか。
なので、このへんをいじった感想を書く。今後も追記するかも。しないかも。
あくまでも自分の場合なので、参考程度に。
・リードを採用する最低カバレッジ数(## 8)
3→10にするとSNP数が0.75倍くらいになった。
思ったよりは減らないんだなー、という印象。
3と10だと質が大きく違う(気がする)ので、わりとお得感があった。
・何個体以上で共通して読めたリードを採用するか(## 12)
全個体数の75%→50%にするとSNP数が1.5倍くらいになった。
このパラメータはSNP数にわりと大きく影響するなー、という印象。
・その他
## 13をいじっても大きくSNP数が変動することはなかった。
## 5はあまりいじってない。それよりはFASTX-toolkitでの処理のパラメータいじったほうがいいかも。
FASTX-toolkitで後ろから削ってやると、削らない場合よりもSNP数が多いという結果に。うまく削ってやると1.3倍くらいになった。
PATHを通す方法|mac OS Xの場合
PATHを通すとは?
こちらを参照に。
「PATHを通す」ってどういうこと? - マクロ生物学徒の備忘録
流れ
ざっくりとした流れは以下の通りである。
・パスを通すべきディレクトリの場所を取得する
・文を付け加えて上書き保存
・再ログインする
順を追って解説する
パスを通すべきディレクトリの場所を取得する
まず、コマンドの意味が書いてあるファイルを探す。
コマンドの意味が書いてあるファイルは、Finderでみると黒い四角に緑の文字で「exec」と書かれたファイル。
ファイル名がそのままターミナルで打ち込むコマンドになる。拡張子はない。
インストールしたソフトウェアのディレクトリの中に[bin]という名前のディレクトリがあったら、おそらくその中にあるはず。
そのファイルがあるディレクトリにパスを通さなければならない。
たとえば、
【フォルダ1】の中に【フォルダ2】があって、
【フォルダ2】の中にコマンドのファイルがある場合、
必ず【フォルダ2】にパスを通す必要がある。
【フォルダ1】にパスを通しても、コマンドは使えるようにならない。
パスを通すべきディレクトリが見つかったら、その場所の「住所」を取得する必要がある。
一番、確実なのは、ターミナルでそのディレクトリに移動して、
pwdと打ち込むと「住所」が表示される。
この「住所」コピーしよう。
これで、パスを通すべきディレクトリの場所を取得できた。
.bash_profileというファイルは、ユーザーログインの時にコンピューターが参照するファイル。
ログイン時にコンピューターがこのファイルをみて、「あ、ここにパスを通せばいいのね」と判断してくれる。
ホームディレクトリにあるが、隠しファイルになっているので、普通FInderでは見えない。
※もし、なければ作る必要がある。ファイル名は必ず「.bash_profile」にすること。
ここでは、デフォルトで入っているテキストエディタのnanoを使って、ターミナル上で開くコマンドを紹介する。
nano ~/.bash_profile※もし、なくても勝手に作ってくれる
文を付け加えて上書き保存
nanoは直感的に操作できるはず。
場所は基本的にはどこでもいいので、以下の一文を付け加えよう。
PATH=$PATH:"Users/○○/△△/□□"※Users/○○/△△/□□は、さっきコピーした「住所」
おわったら
[control]+x
保存するか聞かれるので
y
最後に
[Enter]
を押しておしまい。
打ち込んだ文の意味は
PATHという変数に、もともとPATHに入っていたものと、"Users/○○/△△/□□"をくっつけたものを代入しろ
という意味になる。
例えば、もともと/usr/local/binにパスが通っていた場合、
PATH = "/usr/local/bin" + "Users/○○/△△/□□"
という感じ。
ちなみに、
PATH=$PATH:"Users/○○/△△/□□"の代わりに
PATH="Users/○○/△△/□□":$PATHとやってもほとんど問題ないが、地味に違う。
何が違うかというとパスの優先度が違ってくる。
このへんは、また後日。
再ログイン
画面左上のAppleマークからログアウト・再ログインする。
再起動する必要はない。
再ログインしたら、ちゃんとパスが通っているか確認しよう。
echo $PATHで、さっき追加した「住所」が表示されれば成功。
おわりに
通常はインストール時にデフォルトでパスが通っているディレクトリに実行ファイルをコピーしているので、インストールのたびにそこにパスを通したりする必要はない。
ただ、インストールする権限がないとき、こちらでパスを通してやる必要が出てくる。
そういうときは、自由にいじれるところにインストール用のディレクトリを作り、毎回そのディレクトリを指定してインストールしてやればよい。
そうすれば、パスを通すのは最初の1回だけで、2回め以降はパスを通す過程を省けるのでとてもラク。
ディレクトリを指定してインストールする方法は、また後日。